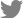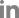ņä£ļĪĀ
ņ¦łļ│æņØä ņ¦äļŗ©ĒĢśĻ│Ā ĒÖśņ×ÉņØś ļ│æļ”¼ņāüĒā£ļź╝ ĒīīņĢģĒĢśņŚ¼ ņĄ£ņĀüņØś ņ╣śļŻīļ▓ĢņØä ņĀ£ņŗ£ĒĢśĻ▒░ļéś ņ╣śļŻīĻ░Ć ņלļÉśĻ│Ā ņ׳ļŖöņ¦Ć ĒīÉļŗ©ĒĢśĻĖ░ ņ£äĒĢ┤, ļŗżņ¢æĒĢ£ ņĪ░ņ¦ü ļ░Å ņāØņ▓┤ņĢĪ(ĒśłņĢĪ, ņÜö, ņ▓ÖņłśņĢĪ, ļ│Ąņłś ļō▒)ņŚÉ ļīĆĒĢ£ ļ®ĆĒŗ░ņśżļ»╣ņŖż(ņ£ĀņĀäņ▓┤, ņĀäņé¼ņ▓┤, Ēøäņä▒ņ£ĀņĀäņ▓┤, ļŗ©ļ░▒ņ▓┤, ņłśņŗØĒÖöļŗ©ļ░▒ņ▓┤, ļīĆņé¼ņ▓┤ ļō▒) ļŹ░ņØ┤Ēä░Ļ░Ć ņāØņé░ļÉśĻ│Ā ņ׳ļŗż. ņ£ĀņĀäņ▓┤, ņĀäņé¼ņ▓┤ļŖö ļČäņäØņØś ņÜ®ņØ┤ņä▒ņ£╝ļĪ£ ņØĖĒĢ┤ Ļ░Ćņן ļ¦ÄņØ┤ ņāØņé░ļÉśņ¢┤ ņÖöņ£╝ļéś, ņŗżņĀ£ ņāØļ¬ģĒśäņāüņŚÉņä£ ņ×æņÜ®ĒĢśļŖö ļŗ©ļ░▒ņ▓┤ ļ░Å ņłśņŗØĒÖöļŗ©ļ░▒ņ▓┤(ņØĖņé░ĒÖöļŗ©ļ░▒ņ▓┤, ļŗ╣ņćäĒÖöļŗ©ļ░▒ņ▓┤, ņĢäņäĖĒŗĖĒÖöļŗ©ļ░▒ņ▓┤, ņ£Āļ╣äĒĆ┤Ēŗ┤ĒÖöļŗ©ļ░▒ņ▓┤ ļō▒)ļź╝ ĻĖ░ļ░śņ£╝ļĪ£ ņ£ĀņĀäņ▓┤, ņĀäņé¼ņ▓┤, ļīĆņé¼ņ▓┤ ļō▒ņØä ĒåĄĒĢ® ļČäņäØĒĢśļŖö ļ░®Ē¢źņ£╝ļĪ£ ņĀäĒÖśņØ┤ ņØ╝ņ¢┤ļéśĻ│Ā ņ׳ļŗż. ĒĢÖĻ│äņŚÉņä£ļŖö CPTAC (Clinical Pro-teomic Tumor Analysis Consortium) [1-3]ņÖĆ ICPC (In-ternational Cancer Proteogenome Consortium) [4,5]Ļ░Ć ļŗ©ļ░▒ņ£ĀņĀäņ▓┤ ņŚ░ĻĄ¼ļōżļĪ£ ņØ┤ļ¤░ ĒŖĖļĀīļō£ļź╝ ņØ┤ļüīĻ│Ā ņ׳Ļ│Ā, ņé░ņŚģĻ│äņŚÉņä£ļŖö ļ¦ÄņØĆ ĒÜīņé¼ļōżņØ┤ ļŗ©ļ░▒ņ▓┤ ĻĖ░ļ░ś ļ®ĆĒŗ░ņśżļ»╣ņŖż ļČäņäØņØä ĒåĄĒĢ£ ņ¦łļ│æ ņ¦äļŗ©, ņ╣śļŻīņĄ£ņĀüĒÖö, ņ╣śļŻīļ¬©ļŗłĒä░ļ¦ü ĻĖ░ņłĀ Ļ░£ļ░£ņŚÉ ļ░Ģņ░©ļź╝ Ļ░ĆĒĢśĻ│Ā ņ׳ļŗż. ņØ┤ļ¤¼ĒĢ£ ņŚ░ĻĄ¼ļōżņØĆ ļ®ĆĒŗ░ņśżļ»╣ņŖż ļŹ░ņØ┤Ēä░ļź╝ ĒåĄĒĢ® ļČäņäØĒĢśņŚ¼ ļ®ĆĒŗ░ņśżļ»╣ņŖż ļČäņ×É ņŗ£ĻĘĖļŗłņ▓śļź╝ ĻĖ░ļ░śņ£╝ļĪ£ ĒÖśņ×É ĻĘĖļŻ╣ņØä ņĀĢņØśĒĢśĻ│Ā, Ļ░ü ĻĘĖļŻ╣ņŚÉ ņĀüĒĢ®ĒĢ£ ņ╣śļŻīļ▓ĢņØä ņĀ£ņŗ£ĒĢśļŖö ĻĘĖļŻ╣ļĀłļ▓©ņØś ņĀĢļ░ĆņØśļŻī ļ░®ļ▓ĢļĪĀņØä ņĀ£ņŗ£ĒĢśĻ│Ā ņ׳ļŗż. ĒĢśņ¦Ćļ¦ī ņ╣śļŻī ņĀäņŚÉ ņłśņ¦æļÉ£ ņŗ£ļŻīņŚÉ ļīĆĒĢ┤ ņāØņé░ļÉ£ ļ®ĆĒŗ░ņśżļ»╣ņŖż ļŹ░ņØ┤Ēä░ļĪ£ļČĆĒä░ ņ╣śļŻīļź╝ ļ░øņ£╝ļ®┤ņä£ ņØ┤ņ¦łņĀüņ£╝ļĪ£ ļ│ĆĒÖöĒĢśļŖö ņ¦łĒÖś ņāüĒā£ņŚÉ ĻĖ░ļ░śĒĢ£ ņĀäņØ┤, ņ×¼ļ░£, ņĢĮļ¼╝ ļ░śņØæņä▒ ļō▒ņŚÉ ļīĆĒĢ£ ņ¦łĒÖś Ēæ£ĒśäĒśĢņØä ņśłņĖĪĒĢśļŖö Ļ▓āņØĆ ĒĢ£Ļ│äĻ░Ć ņ׳ļŗż. ņØ┤ļź╝ ĒĢ┤Ļ▓░ĒĢśĻĖ░ ņ£äĒĢ┤, ņ╣śļŻī ņżæņŚÉļÅä ņłśņ¦æļÉ£ ņŗ£Ļ│äņŚ┤ ņ×äņāüņĀĢļ│┤ļź╝ ļöźļ¤¼ļŗØĒĢśņŚ¼ Ļ░£ņØĖļ│ä ņ¦łĒÖś ņāüĒā£ņØś ņØ┤ņ¦łņä▒ņØä Ļ│ĀļĀżĒĢ£ ņ╣śļŻīņĀ£ļź╝ ņĀ£ņŗ£ĒĢĀ ņłś ņ׳ļŖö Ļ░£ņØĖļ│ä ļ¦×ņČżĒśĢ ņĀĢļ░ĆņØśļŻī ļ░®ļ▓ĢļĪĀņØ┤ Ļ░£ļ░£ļÉśĻ│Ā ņ׳ļŗż. ļ│Ė ļģ╝ļ¼ĖņŚÉņä£ļŖö ņ×äņāüņĀĢļ│┤ ĻĖ░ļ░ś ņĀĢļ░ĆņØśļŻīņŚÉ ļīĆĒĢ┤ņä£ļŖö 2ĒśĢļŗ╣ļć©ļ│æņØä ļīĆņāüņ£╝ļĪ£ ĒĢ£ ļöźļ¤¼ļŗØ ļ¬©ļŹĖ ļČäņäØĻ▓░Ļ│╝ļź╝, ļ®ĆĒŗ░ņśżļ»╣ņŖż ĻĖ░ļ░ś ņĀĢļ░ĆņØśļŻīņŚÉ ļīĆĒĢ┤ņä£ļŖö ņĪ░ĻĖ░ļ░£ļ│æ ņ£äņĢöņŚÉ ļīĆĒĢ£ ļŗ©ļ░▒ņ£ĀņĀäņ▓┤ ļČäņäØĻ▓░Ļ│╝ļź╝ ņśłņŗ£ļĪ£ Ļ░ü ļ░®ļ▓ĢļĪĀņØä ņåīĻ░£ĒĢśĻ│Āņ×É ĒĢ£ļŗż.
ļ│ĖļĪĀ
1. ņ×äņāüņĀĢļ│┤ ĻĖ░ļ░ś ņĀĢļ░ĆņØśļŻīņØś ņśł
ļ©╝ņĀĆ ņ¦Ćļé£ 20ļģäĻ░ä ņä£ņÜĖļīĆĒĢÖĻĄÉļ│æņøÉĻ│╝ ļČäļŗ╣ņä£ņÜĖļīĆĒĢÖĻĄÉļ│æņøÉ ļé┤ļČäļ╣äļé┤Ļ│╝ļź╝ ļé┤ņøÉĒĢ£ 18ļ¦ī ļ¬ģņØś ļŗ╣ļć©ļ│æĒÖśņ×É ņĮöĒśĖĒŖĖņŚÉ ļīĆĒĢ┤ņä£ ņ¢╗ņ¢┤ņ¦ä ĻĖ░ļ│Ė ņ×äņāüņĀĢļ│┤, ĒśłņĢĪ Ļ▓Ćņé¼ ļŹ░ņØ┤Ēä░(CBC [complete blood count] ļŹ░ņØ┤Ēä░ ĒżĒĢ©), ņÜöļČäņäØ ļŹ░ņØ┤Ēä░, Ēł¼ņĢĮ ņĀĢļ│┤ ļō▒ņØś ņóģņåŹļŹ░ņØ┤Ēä░ļź╝ ņłśņ¦æĒĢśņśĆļŗż. ņłśņ¦æļÉ£ ņŗ£Ļ│äņŚ┤ ņ×äņāüņĀĢļ│┤ ļŹ░ņØ┤Ēä░ļōżņØä ņŚ░ņåŹļ│Ćņłś(ņśł: alanine aminotransferase [ALT], ņĮ£ļĀłņŖżĒģīļĪż ņłśņ╣ś; Fig. 1A, ļģ╣ņāē ļ░ĢņŖż), ņØ┤ņé░ļ│Ćņłś(ņśł: Ēł¼ņĢĮļÉ£ ņĢĮļ¼╝, ļéśņØ┤, ņä▒ļ│ä ļō▒; Fig. 1A, Ēīīļ×Ćņāē ļ░ĢņŖż)ļĪ£ ļéśļłäĻ│Ā, Ļ░ü ļ│Ćņłś ĻĘĖļŻ╣ņŚÉ ļīĆĒĢ┤ņä£ ņł£ĒÖś ņŗĀĻ▓Įļ¦Ø(recurrent neural network, RNN)ņØä ĻĄ¼ņČĢĒĢśĻ│Ā, ņØ┤ļōżņØä ļŗżņĖĄ ĒŹ╝ņģēĒŖĖļĪĀ(multilayer perceptron, MLP)ņØä ņØ┤ņÜ®ĒĢśņŚ¼ ĒåĄĒĢ®ĒĢśļŖö ļöźļ¤¼ļŗØ ļ¬©ļŹĖņØä ĻĄ¼ņČĢĒĢśņśĆļŗż. ņØ┤ļź╝ ņØ┤ņÜ®ĒĢśņŚ¼ ļŗ╣ĒÖöĒśłņāēņåī(HbA1c)ņÖĆ ņČöņĀĢņé¼ĻĄ¼ņ▓┤ņŚ¼Ļ│╝ņ£©(estimated glomerular filtration rate, eGFR)ņØä ļ¦żĒĢæĒĢśņśĆļŗż. RNN ĒĢÖņŖĄ Ļ▓░Ļ│╝ ļŗ╣ĒÖöĒśłņāēņåīņŚÉ ļīĆĒĢ┤ņä£ļŖö R2 = 0.75, eGFRņŚÉ ļīĆĒĢ┤ņä£ļŖö R2 = 0.97ņØ┤ņŚłļŗż(Fig. 1B). ņØ┤ļĢī MLP ņ▓½ ļ▓łņ¦Ė ļĀłņØ┤ņ¢┤ņŚÉ ņ×äļ▓Āļö®ļÉ£ ĒÖśņ×ÉļōżņØś ņ£Āņé¼ņä▒ņØä ņØ┤ņÜ®ĒĢśņŚ¼ 7Ļ░£ņØś ĒÖśņ×É ĻĘĖļŻ╣(Cluster 0Ōł╝6)ņØä ļÅÖņĀĢĒĢśņśĆļŗż(Fig. 1C). ņØ┤ļōż 7Ļ░£ņØś ĻĘĖļŻ╣ ņżæņŚÉņä£ ļŗ╣ĒÖöĒśłņāēņåīĻ░Ć ņāüņŖ╣ĒĢśļŖö Cluster 0, 1, 4, 6 (Fig. 1D)ņŚÉ ĒŖ╣ņØ┤ņĀüņ£╝ļĪ£ ņ”ØĻ░ĆĒĢ£ ļ│ĆņłśļōżņØä ļÅÖņĀĢĒĢśņŚ¼, Ļ░äņåÉņāü ņłśņ╣śņØĖ ALT ļĀłļ▓©ņØ┤ ņ”ØĻ░ĆĒĢ£ Cluster 1ņØĆ metabolism-mediated insulin resistance, Cļ░śņØæļŗ©ļ░▒ņ¦ł(C-reactive protein)ņØ┤ ņ”ØĻ░ĆĒĢ£ Cluster 4ļŖö inflammation-mediated insulin resistance, ĒśĖļź┤ļ¬¼ ņłśņ╣ś(thyroid stimulating hormone)Ļ░Ć ņ”ØĻ░ĆĒĢ£ Cluster 6ņØĆ hormone-mediated insulin resistance ļź╝ Ļ░Ćņ¦ĆļŖö ĒÖśņ×É ĻĘĖļŻ╣ņ£╝ļĪ£ ĒīīņĢģļÉśņŚłļŗż(Fig. 1E). ļŗżļ¦ī Cluster 0ņŚÉņä£ ņ”ØĻ░ĆĒĢ£ ĒśĖņŚ╝ĻĖ░ĻĄ¼(basophil)ņÖĆ ņØĖņŖÉļ”░ņĀĆĒĢŁņä▒Ļ│╝ņØś Ļ┤ĆļĀ©ņä▒ņØĆ ļ¬ģĒÖĢĒĢśņ¦Ć ņĢŖņĢśļŗż. ņØ┤ļōż ĒÖśņ×É ĻĘĖļŻ╣ļ│ä ņØĖņŖÉļ”░ņĀĆĒĢŁņä▒ņŚÉ ļīĆĒĢ£ ņØ┤ĒĢ┤ļź╝ ĻĖ░ļ░śņ£╝ļĪ£ ĻĘĖļŻ╣ļ│äļĪ£ ņ╣śļŻīņĀ£ļź╝ ņĀ£ņŗ£ĒĢĀ ņłś ņ׳ļŗż. ņśłļź╝ ļōżņ¢┤, ņŚ╝ņ”ØņØ┤ ņ”ØĻ░ĆĒĢ£ Cluster 4ņŚÉ ļīĆĒĢ┤ņä£ļŖö ņŚ╝ņ”Ø ņ¢ĄņĀ£ņĀ£ņÖĆ ļŗ╣ļć©ļ│æņĢĮņØä Ļ░ÖņØ┤ ņ▓śļ░®ĒĢĀ ņłś ņ׳ļŗż.
Fig.┬Ā1.
RNN (recurrent neural network)-based deep learning model using time-course clinical data. (A) Deep learning model architecture. X i,j indicates clinical data measured or drug types (i) used at time j. Forty clinical variables and 20 drug types were used. The other numbers indicate the node numbers in the corresponding neural networks. (B) Prediction accuracy of the deep learning model. (C) Patient clustering using the embedded states in the 1st multilayer perceptron (MLP) layer. (D) Distribution of temporal profiles of the measured (True) and predicted glycated hemoglobin HbA1c (Predicted) in patients included in each cluster. Principal component analysis (PCA) plot and distance represent how the embedded state in the 1st MLP layer vary over time. Blue to yellow colors in PCA plot represent early to late time points, respectively. (E) Contribution of each clinical variables and drug types to each cluster. T values indicate positive and negative contributions to defining each cluster. Clinical variables highly contributing to individual clusters are indicated in red. (F) Actual measured (blue) and model predicted (orange) glycated hemoglobin changes over years and actual prescribed (blue) and model determined (orange) treatment regimens.
eGFR, estimated glomerular filtration rate; LSTM, long short-term memory; UMAP, uniform manifold approximation and projection.
ļ®ĆĒŗ░ņśżļ»╣ņŖżņÖĆ ļŗ¼ļ”¼ RNN ĻĖ░ļ░ś ļöźļ¤¼ļŗØ ļ¬©ļŹĖņØĆ Ļ░£ļ│ä ĒÖśņ×Éļ│äļĪ£ ņ╣śļŻīņĀ£ņÖĆ ļŗ╣ĒÖöĒśłņāēņåī, eGFRņØä ļ¦żĒĢæĒĢĀ ņłś ņ׳ņ£╝ļ»ĆļĪ£, ņāłļĪ£ņÜ┤ ĒÖśņ×ÉņŚÉ ļīĆĒĢ┤ņä£ļÅä ĒĢ┤ļŗ╣ ĒÖśņ×ÉņØś ņ×äņāüņĀĢļ│┤, ĒśłņĢĪŃåŹņÜö ļČäņäØ ļŹ░ņØ┤Ēä░ļź╝ ņ¢╗ņØĆ Ēøä ļ¬©ļōĀ Ļ░ĆņāüņØś Ēł¼ņĢĮ ņĀĢļ│┤(ņ¦Ćļé£ 20ļģäĻ░ä ĒĢ£ ļ▓łņØ┤ļØ╝ļÅä ņ▓śļ░®ļÉ£ Ēł¼ņĢĮļ▓Ģ)ļź╝ ļöźļ¤¼ļŗØ ļ¬©ļŹĖņŚÉ ļäŻĻ│Ā ļŗ╣ĒÖöĒśłņāēņåīļź╝ ņśłņĖĪĒĢ£ ļÆż, Ļ░Ćņן ļŗ╣ĒÖöĒśłņāēņåī Ļ░Éņåīļź╝ ļ│┤ņØ┤ļ®┤ņä£ eGFR ņČöņĀĢņ╣śļŖö ņĢłņĀĢļÉ£ ĻĄ¼Ļ░äņŚÉ ņåŹĒĢśļŖö Ēł¼ņĢĮļ▓ĢņØä ĒÖśņ×ÉņŚÉĻ▓ī Ēł¼ņĢĮĒĢ┤ņĢ╝ ĒĢśļŖö ņĄ£ņĀüņØś ņ╣śļŻīņĀ£ļĪ£ ņäĀņĀĢĒĢśņśĆļŗż(Fig. 1F, ņśżļźĖņ¬Į Ēæ£ - Ēīīļ×Ćņāē: ņŗżņĀ£ Ēł¼ņĢĮļÉ£ ņĢĮļ¼╝, ņśżļĀīņ¦Ćņāē: ļ¬©ļŹĖņØ┤ ņĀĢĒĢ£ ņĢĮļ¼╝; ņÖ╝ņ¬Į ĒöäļĪ£ĒīīņØ╝ - Ēīīļ×Ćņāē: ņŗżņĀ£ ļŗ╣ĒÖöĒśłņāēņåī ļ│ĆĒÖö, ņśżļĀīņ¦Ćņāē: ļ¬©ļŹĖņØ┤ ņĀĢĒĢ£ ņĢĮļ¼╝ņØ┤ ņ▓śļ░®ļÉśņŚłņØä ļĢī ļ¬©ļŹĖņØ┤ ņśłņĖĪĒĢ£ ļŗ╣ĒÖöĒśłņāēņåī ļ│ĆĒÖö). ņØ┤ļ¤¼ĒĢ£ ļöźļ¤¼ļŗØ ļ¬©ļŹĖņØä ĒåĄĒĢ£ ņĄ£ņĀüņØś ņ╣śļŻīņĀ£ņØś ņäĀņĀĢņØĆ ņĢ×ņ£╝ļĪ£ ļŗ╣ļć©ļ│æĒÖśņ×ÉņØś ņ╣śļŻīņĀ£ ņäĀĒāØ ņŗ£ ĻĖ░ļ│ĖņĀüņ£╝ļĪ£ Ļ│ĀļĀżĒ¢łļŹś ņ×äņāüņāüĒÖ®(ļÅÖļ░śņ¦łĒÖś, ņĀĆĒśłļŗ╣ ņ£äĒŚśļÅä, ļČĆņ×æņÜ®, ļéśņØ┤, ļ╣äņÜ® ļō▒)ņŚÉ ļŹöĒĢśņŚ¼ ĒĢśļéśņØś ĻĘ╝Ļ▒░ļĪ£ ņé¼ņÜ®ļÉĀ ņłś ņ׳Ļ│Ā ļŗ╣ļć©ļ│æ ņ╣śļŻīņŚÉņä£ņØś ņĀĢļ░ĆņØśļŻīņŚÉ ņóĆ ļŹö ĻĘ╝ņĀæĒĢśļŖö ļ░£ĒīÉņØ┤ ļÉĀ ņłś ņ׳Ļ▓Āļŗż.
2. ļ®ĆĒŗ░ņśżļ»╣ņŖż ĻĖ░ļ░ś ņĀĢļ░ĆņØśļŻīņØś ņśł
80ļ¬ģņØś ņĪ░ĻĖ░ļ░£ļ│æ ņ£äņĢöĒÖśņ×ÉļĪ£ļČĆĒä░ ņ£äņĢö ņĪ░ņ¦üĻ│╝ ņŻ╝ņ£ä ņĀĢņāü ņĪ░ņ¦ü ņŗ£ļŻīņŚÉ ļīĆĒĢ┤ ņĀäņé¼ņ▓┤, ĻĖĆļĪ£ļ▓īļŗ©ļ░▒ņ▓┤, ņØĖņé░ĒÖöļŗ©ļ░▒ņ▓┤, ļŗ╣ņćäĒÖöļŗ©ļ░▒ņ▓┤ ļČäņäØņØä ĒåĄĒĢ┤ ņĀäļĀ╣RNA (messenger RNA, mRNA) ļ░£Ēśäļ¤ē, ļŗ©ļ░▒ņ¦ł ļ░£Ēśäļ¤ē, ņØĖņé░ĒÖö ņĀĢļÅä, ļŗ╣ņćäĒÖö ņĀĢļ│┤ ļō▒ņØś ļŹ░ņØ┤Ēä░ļź╝ ņāØņé░ĒĢśņśĆļŗż. Ļ░ü ņśżļ»╣ņŖż ļŹ░ņØ┤Ēä░ņŚÉņä£ median absolute deviation ņāüņ£ä 10, 20, 30 ļ░▒ļČäņ£äņŚÉ ņåŹĒĢśļŖö ņ£ĀņĀäņ×É, ļŗ©ļ░▒ņ¦ł, ņØĖņé░ĒÖö, ļŗ╣ņćäĒÖö ļŗ©ļ░▒ņ¦łņØä ņäĀņĀĢĒĢśĻ│Ā, ņØ┤ļōżņØä ņØ┤ņÜ®ĒĢśņŚ¼ non-negative matrix factorization Ēü┤ļ¤¼ņŖżĒä░ļ¦üņØä ņłśĒ¢ēĒĢśņśĆļŗż. Ēü┤ļ¤¼ņŖżĒä░ļ¦ü ļ░śļ│Ą ņłśĒ¢ēņŚÉņä£ņØś ņ×¼Ēśäņä▒ņØä consensus value matrixļĪ£ Ēæ£ĒśäĒĢśĻ│Ā, ņØ┤ matrixņŚÉ ļīĆĒĢ£ Ēü┤ļ¤¼ņŖżĒä░ļ¦üņØä ņłśĒ¢ē(Fig. 2A-D, ņÖ╝ņ¬Į heat map)ĒĢśņŚ¼ cophenetic cor-relationņØä Ļ│äņé░ĒĢśņśĆļŗż. Cophenetic correlationņØś Ēü┤ļ¤¼ņŖżĒä░ Ļ░£ņłśņŚÉ ļö░ļźĖ ļ│ĆĒÖöļź╝ Ļ┤Ćņ░░ĒĢśņŚ¼ Ēü┤ļ¤¼ņŖżĒä░(ņĢäĒśĢ, subtype)ņØś Ļ░£ņłśļź╝ Ļ▓░ņĀĢĒĢśĻ│Ā, ĻĘĖ Ļ░£ņłśņŚÉ ĒĢ┤ļŗ╣ĒĢśļŖö ĒÖśņ×Éļ│ä ņĢäĒśĢņØä ņśłņĖĪĒĢśņśĆļŗż. Ļ░ü ņśżļ»╣ņŖż ļŹ░ņØ┤Ēä░ļĪ£ļČĆĒä░ ņśłņĖĪļÉ£ ĒÖśņ×É ņĢäĒśĢņŚÉ ņåŹĒĢśļŖöņ¦Ć ņŚ¼ļČĆļź╝ binary value matrixļĪ£ ļ¦īļōĀ Ēøä, Ļ░ü ļŹ░ņØ┤Ēä░ļĪ£ļČĆĒä░ ņśłņĖĪļÉ£ ņĢäĒśĢņØ┤ ņä£ļĪ£ ņØ╝ņ╣śĒĢśļŖö ņĀĢļÅäļź╝ Ļ│ĀļĀżĒĢśņŚ¼ ņĄ£ņóģ ĒÖśņ×É ņĢäĒśĢņØś Ļ░£ņłśļź╝ 4Ļ░£(Sub1Ōł╝4)ļĪ£ Ļ▓░ņĀĢĒĢśņśĆļŗż(Fig. 2E). Ļ░ü Ēü┤ļ¤¼ņŖżĒä░ņØś ĒŖ╣ņä▒ņØä ņØ┤ĒĢ┤ĒĢśĻĖ░ ņ£äĒĢ┤ Ļ░ü ņśżļ»╣ņŖż ļŹ░ņØ┤Ēä░ļĪ£ļČĆĒä░ ņśłņĖĪļÉ£ ņĢäĒśĢļ│ä ĒÖśņ×Éļōż Ļ░äņØś ļ░£Ēśäļ¤ē ļ╣äĻĄÉļČäņäØņØä ņłśĒ¢ēĒĢ©ņ£╝ļĪ£ņŹ© Ļ░ü ņĢäĒśĢņŚÉņä£ ļéśļ©Ėņ¦Ć ņĢäĒśĢļōż(ņśł: Prot1 vs. Prot2Ōł╝4)ņŚÉ ļ╣äĒĢ┤ ļ░£Ēśäļ¤ēņØ┤ ņ£ĀņØśĒĢśĻ▓ī ļåÆņØĆ(P < 0.05) mRNA, ļŗ©ļ░▒ņ¦ł, ņØĖņé░ĒÖö ņŗ£ĻĘĖļŗłņ▓śļōżņØä ļÅÖņĀĢĒĢśņśĆļŗż.
Fig.┬Ā2.
Determination and characterization of subtypes of patients with early-onset gastric cancers through integrative clustering using mRNA and protein data (A-D). (A) mRNA, RNA1Ōł╝2. (B) Protein, Prot1Ōł╝4. (C) Phosphorylation, Phos1Ōł╝3. (D) N-glycosylation, Gly1Ōł╝3. For example, RNA1 was defined by upregulation of 919 genes (rna1). (E) Four subtypes (Sub1Ōł╝4) determined by integrated clustering of individual omics-based clusters. (F) Cellular pathways enriched by the mRNA (rna1Ōł╝2) or protein signatures (prot1Ōł╝4, phos1Ōł╝3, or gly1Ōł╝3) defining each subtype. Colors in the heat map indicate the enrichment significance, -log10(P value), and the pathways with P Ōēż 0.05 were denoted in color (see the color bar).
Adapted from the article of Mun et al. (Cancer Cell 2019;35:111-24.e10) [4] with original copyright holder's permission.
ņØ┤ļōż ņŗ£ĻĘĖļŗłņ▓śļōż(Fig. 2A-D, ņśżļźĖņ¬Į rna1Ōł╝2, prot1Ōł╝4, phos1Ōł╝3, gly1Ōł╝3)ņŚÉ ļīĆĒĢ£ ConsensusPathDBļź╝ ņØ┤ņÜ®ĒĢśņŚ¼ gene set enrichment analysis (GSEA)ļź╝ ņłśĒ¢ēĒĢ©ņ£╝ļĪ£ņŹ©, Ļ░ü ņĢäĒśĢĻ│╝ ĒåĄĻ│äņĀüņ£╝ļĪ£ ņ£ĀņØśĒĢśĻ▓ī Ļ┤ĆļĀ©ļÉ£ ņäĖĒżĻ▓ĮļĪ£ļź╝ ļÅÖņĀĢĒĢśņśĆļŗż(Fig. 2F). mRNA, ļŗ©ļ░▒ņ¦ł ņŗ£ĻĘĖļŗłņ▓śļōżņØ┤ ņäĖĒżļČäĒÖö(cell proliferation) Ļ┤ĆļĀ© ņäĖĒżĻ▓ĮļĪ£ļź╝ ļīĆļ│ĆĒĢśļ»ĆļĪ£ Sub1ņØä prolif-erative tumor; mRNA, ļŗ©ļ░▒ņ¦ł ņŗ£ĻĘĖļŗłņ▓śļōżņØ┤ ļ®┤ņŚŁ Ļ┤ĆļĀ© Ļ▓ĮļĪ£ļź╝ ļīĆļ│ĆĒĢśļ»ĆļĪ£ Sub2ļź╝ immunogenic tumor; ļŗ©ļ░▒ņ¦ł ņŗ£ĻĘĖļŗłņ▓śļōżņØ┤ ļīĆņé¼Ļ┤ĆļĀ© Ļ▓ĮļĪ£ļź╝ ļīĆļ│ĆĒĢśļ»ĆļĪ£ Sub3ņØä metabolic tumor; mRNA, ļŗ©ļ░▒ņ¦ł ņŗ£ĻĘĖļŗłņ▓śļōżņØ┤ invasion ļ░Å epithe-lial-mesenchymal transition (EMT) Ļ┤ĆļĀ© Ļ▓ĮļĪ£ļź╝ ļīĆļ│ĆĒĢśļ»ĆļĪ£ Sub4ļź╝ invasive tumorļĪ£ ņĀĢņØśĒĢśņśĆļŗż.
Ļ░ü ņĢäĒśĢļ│ä ņĢöņØś ĻĖ░ļŖźņĀü ĒŖ╣ņä▒ņØä ļīĆļ│ĆĒĢśļŖö ņäĖĒżĻ▓ĮļĪ£ņŚÉ ņåŹĒĢśļŖö mRNA, ļŗ©ļ░▒ņ¦ł, ņØĖņé░ĒÖö ņŗ£ĻĘĖļŗłņ▓śļź╝ ņäĀļ│äĒĢśĻ│Ā, Ļ│ĄņÜ® ļŹ░ņØ┤Ēä░ļ▓ĀņØ┤ņŖżņāüņØś ļŗ©ļ░▒ņ¦ł-ļŗ©ļ░▒ņ¦ł ņāüĒśĖņ×æņÜ® ņĀĢļ│┤, Ēī©ņŖżņø©ņØ┤ DB ļ░Å ĻĖ░ņĪ┤ ļ¼ĖĒŚīņāüņØś ņĪ░ņĀłĻ┤ĆĻ│ä ņĀĢļ│┤ļź╝ ļ░öĒāĢņ£╝ļĪ£ ņäĀļ│äļÉ£ ņĢäĒśĢļ│ä ņŗ£ĻĘĖļŗłņ▓ś ļ░Å ĻĘĖļōżĻ│╝ ņāüĒśĖņ×æņÜ®ĒĢśļŖö ļČäņ×ÉļōżļĪ£ ĻĄ¼ņä▒ļÉ£ ņĢäĒśĢļ│ä ļäżĒŖĖņøīĒü¼ ļ¬©ļŹĖņØä ĻĄ¼ņČĢĒĢśņśĆļŗż(Fig. 3). Sub2 immunogenic tumorļź╝ ļīĆļ│ĆĒĢśļŖö ļäżĒŖĖņøīĒü¼ ļ¬©ļŹĖņØĆ ņĢöņØ┤ ļČäļ╣äĒĢśļŖö ņĢöĒĢŁņøÉņØä antigen presenting (AP) cellņØ┤ ņŗØĻĘĀ ņ×æņÜ®(phagocyto-sis)ņØä ĒĢśĻ│Ā, ņØ┤ļź╝ ĒĢŁņøÉņ▓śļ”¼(processing)ĒĢśņŚ¼ ņĢöĒĢŁņøÉņØä ĒĢŁņøÉņĀ£ņŗ£(presentation)ĒĢśļ®┤, cytotoxic T cellņØ┤ ņØ┤ļź╝ ņØĖņ¦ĆĒĢśņŚ¼ ņĢöņäĖĒżļź╝ ņŻĮņØ┤ļŖö ĒĢŁņĢöļ®┤ņŚŁ(anti-tumor immunity)ņØä ļ│┤ņŚ¼ņŻ╝Ļ│Ā ņ׳ļŗż(Fig. 3A). Sub4 invasive tumorļź╝ ļīĆļ│ĆĒĢśļŖö ļäżĒŖĖņøīĒü¼ ļ¬©ļŹĖņØĆ ņ£äņĢöņŚÉņä£ invasionĻ│╝ EMTņŚÉ ņżæņÜöĒĢśļŗżĻ│Ā ņĢīļĀżņ¦ä RhoA ņŗĀĒśĖĻ▓ĮļĪ£ņØś upstream, downstream Ļ▓ĮļĪ£ļź╝ ļ│┤ņŚ¼ņŻ╝Ļ│Ā ņ׳ļŗż(Fig. 3B). Sub2, 4 ļäżĒŖĖņøīĒü¼ ļ¬©ļŹĖņŚÉņä£ ļŗ©ļ░▒ņ¦ł ņŗ£ĻĘĖļŗłņ▓ś(ļģĖļō£ ļ│┤ļŹö, P, GļĪ£ Ēæ£ņŗ£ļÉ£ ļģĖļō£ļōż)ļź╝ ņĀ£Ļ▒░ĒĢśļ®┤ ĒĢŁņĢöļ®┤ņŚŁĻ│╝ RhoA Ļ▓ĮļĪ£ļź╝ ņĀ£ļīĆļĪ£ ĒīīņĢģĒĢĀ ņłś ņŚåļŗżļŖö Ļ▓āņØä ņĢī ņłś ņ׳ļŗż.
Fig.┬Ā3.
Network models showing pathways defined by the interactions among the genes (rna2 for Sub2 and rna1 for Sub4) and proteins (prot1, phos3, and gly3 for Sub2 and prot2, phos1, and gly1 for Sub4; see Fig. 2E) predominantly upregulated in Sub2 (A) and Sub4 (B). Node colors and circled labels, respectively, represent upregulation of mRNA (node center), protein (node boundary), phosphorylation (circled P), and glycosylation (circled G) levels in each network. Adapted from the article of Mun et al. (Cancer Cell 2019;35:111-24.e10) [4] with original copyright holder's permission.

ļ¦łņ¦Ćļ¦ēņ£╝ļĪ£ GSEA Ļ▓░Ļ│╝(Fig. 2F)ņÖĆ ļäżĒŖĖņøīĒü¼ ļ¬©ļŹĖņØä ĒåĄĒĢ®ĒĢśļ®┤, Ļ░ü ņĢäĒśĢņŚÉ ņåŹĒĢśļŖö ĒÖśņ×ÉļōżņŚÉ ļīĆĒĢ£ ņ╣śļŻīļ░®ļ▓ĢņØä ņĀ£ņŗ£ĒĢĀ ņłś ņ׳ļŗż. ņśłļź╝ ļōżņ¢┤, Ļ░Ćņן ņśłĒøäĻ░Ć ņĢł ņóŗņØĆ Sub4 invasive tumor ņŚÉ ļīĆĒĢ┤ņä£ļŖö RhoA ņŗĀĒśĖĻ▓ĮļĪ£ļź╝ ņ¢ĄņĀ£ĒĢ┤ņĢ╝ ĒĢśĻ│Ā, GSEA Ļ▓░Ļ│╝ļź╝ ļ│┤ļ®┤ ĒĢŁņĢöļ®┤ņŚŁ(anti-tumor immunity)ņØ┤ ņĀĆĒĢśļÉ£ Ļ▓āņØä ņĢī ņłś ņ׳ļŗż. ņØ┤ņÖĆ Ļ┤ĆļĀ©ļÉ£ Sub4ņØś mRNA, ļŗ©ļ░▒ņ¦ł ņŗ£ĻĘĖļŗłņ▓śļź╝ ļČäņäØĒĢ┤ļ│┤ļ®┤ myeloid-derived suppressive cell (MDSC)ņØ┤ ņ”ØĻ░ĆļÉśņ¢┤ ņ׳Ļ│Ā PVR (PVR cell adhesion molecule)Ļ│╝ Ļ░ÖņØĆ ļ®┤ņŚŁĻ┤Ćļ¼Ė(immune checkpoint)ļōżņØ┤ ņ”ØĻ░ĆļÉśņ¢┤ ņ׳ļŗżļŖö Ļ▓āņØä ņĢī ņłś ņ׳ļŗż. ļö░ļØ╝ņä£, Sub4 ņĢöņØś ņ╣śļŻīņĀ£ļĪ£ RhoA ņ¢ĄņĀ£ņĀ£(ņśł: ROCK inhibitors), MDSC ņ¢ĄņĀ£ņĀ£(ņśł: IL-10), ļ®┤ņŚŁĻ┤Ćļ¼Ėņ¢ĄņĀ£ņĀ£(ņśł: PVRņØä ņ¢ĄņĀ£ĒĢśļŖö TIGIT)ļōżņŚÉ ĻĖ░ļ░śĒĢ£ ļ│æņÜ®ņ╣śļŻīļź╝ ņĄ£ņĀü ņ╣śļŻīņĀ£ļĪ£ ņĀ£ņŗ£ĒĢĀ ņłś ņ׳ļŗż.
Ļ▓░ļĪĀ
ļ®ĆĒŗ░ņśżļ»╣ņŖż ĻĖ░ļ░ś ņĀĢļ░ĆņØśļŻī ĻĖ░ņłĀ Ļ░£ļ░£ņØ┤ ĒÖśņ×ÉņØś ņ¦äļŗ©Ļ│╝ ņ╣śļŻīņŚÉņä£ Ēī©ļ¤¼ļŗżņ×ä ņĀäĒÖś(paradigm shift)ņØä Ļ░ĆņĀĖļŗż ņżä Ļ▓āņØĆ ņé¼ņŗżņØ┤ļéś, ņśżļ»╣ņŖż ļČäņäØņØĆ ĻĖ░ļ│ĖņĀüņ£╝ļĪ£ ļ¦ÄņØĆ ļŹ░ņØ┤Ēä░ ņāØņé░ ļ╣äņÜ®ņØ┤ ļōżņ¢┤ ļ¬©ļōĀ ĒÖśņ×ÉļōżņŚÉ ļīĆĒĢ┤ ļ®ĆĒŗ░ņśżļ»╣ņŖż ļŹ░ņØ┤Ēä░ļź╝ ņāØņé░ĒĢśļŖö Ļ▓āņØĆ ņ¢┤ļĀĄļŗż. CPTAC, ICPCņØś Ļ▓ĮņÜ░ņŚÉļÅä 200ļ¬ģ ņØ┤ĒĢśņØś ĒÖśņ×ÉņŚÉņä£ ļ®ĆĒŗ░ņśżļ»╣ņŖż ļČäņäØņØ┤ ņłśĒ¢ēļÉśņŚłļŗż. ņśżļ»╣ņŖż ļŹ░ņØ┤Ēä░ņÖĆļŖö ļŗ¼ļ”¼, Ēśäņ×¼ ņ×äņāüņŚÉņä£ ĒÖśņ×ÉņØś ņāüĒā£ ĒīīņĢģņØä ņ£äĒĢ┤ ņĖĪņĀĢĒĢśļŖö ĻĖ░ļ│Ė ņĀĢļ│┤(Ēéż, ļ¬Ėļ¼┤Ļ▓ī, ĒśłņĢĢ, ņ▓┤ņ¦łļ¤ēņ¦Ćņłś ļō▒), ĒśłņĢĪŃåŹņÜö Ļ▓Ćņé¼ļź╝ ĒåĄĒĢ£ ņ¦łĒÖśļ│ä ņĀĢļ│┤(ļŗ╣ļć©ļ│æņØś Ļ▓ĮņÜ░ ļŗ╣ĒÖöĒśłņāēņåī, Ēśłļŗ╣, Ēü¼ļĀłņĢäĒŗ░ļŗī, ņĮ£ļĀłņŖżĒģīļĪż ņłśņ╣ś ļō▒), ņśüņāü(ņ╗┤Ēō©Ēä░ļŗ©ņĖĄņ┤¼ņśü, ņ×ÉĻĖ░Ļ│Ąļ¬ģņśüņāü, ņ¢æņĀäņ×Éļŗ©ņĖĄņ┤¼ņśü ļō▒)ņØä ĒżĒĢ©ĒĢśļŖö ņ×äņāüņĀĢļ│┤ļŖö ļ¬©ļōĀ ĒÖśņ×ÉņŚÉ ļīĆĒĢ┤ ņĪ┤ņ×¼ĒĢ£ļŗż. ļö░ļØ╝ņä£ ņØ┤ļōż ņ×äņāüņĀĢļ│┤ ļ╣ģļŹ░ņØ┤Ēä░ņÖĆ ļ®ĆĒŗ░ņśżļ»╣ņŖż ļŹ░ņØ┤Ēä░ļź╝ ĒåĄĒĢ®ĒĢśņŚ¼ Ļ░£ņäĀļÉ£ ņĀĢļ░ĆņØśļŻī ĻĖ░ņłĀņØä Ļ░£ļ░£ĒĢśļŖö ļģĖļĀźļōżņØ┤ ĒÖ£ļ░£ĒĢśĻ▓ī ņ¦äĒ¢ēļÉśĻ│Ā ņ׳ļŗż.



 PDF Links
PDF Links PubReader
PubReader ePub Link
ePub Link Full text via DOI
Full text via DOI Download Citation
Download Citation Print
Print